Capítulo 40 Proyectos reproducibles con RStudio y R Markdown
El producto final de un proyecto de análisis de datos frecuentemente es un informe. Muchas publicaciones científicas pueden considerarse como un informe final de un análisis de datos. Lo mismo es cierto para los artículos de noticias basados en datos, un informe de análisis para su empresa o notas para una clase sobre cómo analizar datos. Los informes suelen estar en papel o en un PDF que incluyen una descripción textual de los resultados junto con algunos gráficos y tablas resultantes del análisis.
Imaginen que después de finalizar el análisis y el informe, les informan que les dieron el set de datos incorrecto, les enviarán uno nuevo y les piden que ejecuten el mismo análisis con este nuevo set de datos. ¿O qué pasa si se dan cuenta de que cometieron un error y necesitan volver a examinar el código, corregir el error y volver a ejecutar el análisis? ¿O supongan que alguien a quien están entrenando quiere ver el código y poder reproducir los resultados para conocer su enfoque?
Situaciones como las que acabamos de describir son bastante comunes para los científicos de datos. Aquí, describimos cómo pueden mantener sus proyectos de ciencia de datos organizados con RStudio para que el proceso de volver a ejecutar un análisis sea sencillo. Luego, demostraremos cómo generar informes reproducibles con R Markdown y el paquete knitR de una manera que ayudará enormemente a recrear informes con esfuerzo mínimo. Esto es posible debido a que los documentos de R Markdown permiten combinar códigos y descripciones textuales en el mismo documento, y las figuras y tablas producidas por el código se agregan automáticamente al documento.
40.1 Proyectos de RStudio
RStudio ofrece una manera de mantener todos los componentes de un proyecto de análisis de datos organizados en una carpeta y mantener un registro de la información sobre este proyecto, como el estatus Git de los archivos, en un archivo. En la Sección 39.6, demostramos cómo RStudio facilita el uso de Git y GitHub a través de proyectos de RStudio. En esta sección, demostramos cómo comenzar un nuevo proyecto y ofrecemos algunas recomendaciones sobre cómo mantenerlo organizado. Los proyectos de RStudio también les permiten tener abiertas varias sesiones de RStudio y mantener un registro de cuál es cuál.
Para comenzar un proyecto, hagan clic en File y luego en New Project. Muchas veces ya hemos creado una carpeta para guardar el trabajo, como lo hicimos en la Sección 38.7, en cual caso seleccionamos Existing Directory. Aquí les mostramos un ejemplo en el que aún no hemos creado una carpeta y seleccionamos la opción New Directory.
Entonces para un proyecto de análisis de datos, generalmente seleccionan la opción New Project:
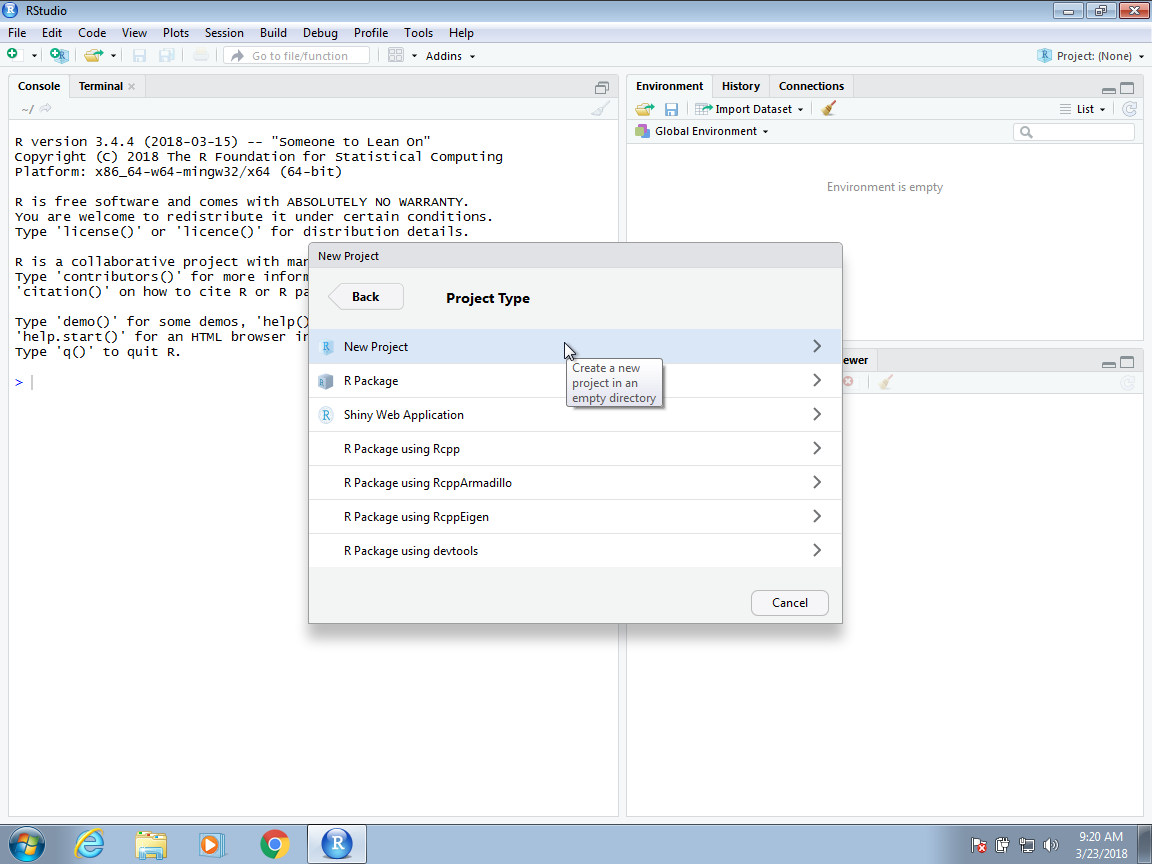
Ahora tendrán que decidir la ubicación de la carpeta que se asociará con su proyecto, así como el nombre de la carpeta. Al igual que con los nombres de archivo, cuando eligen un nombre de carpeta, aasegúrense de que sea un nombre significativo que los ayude a recordar de qué se trata el proyecto. Además, al igual que con los archivos, recomendamos usar letras minúsculas, sin espacios y con guiones para separar las palabras. Llamaremos a la carpeta para este proyecto my-first-project. Esto generará un archivo Rproj llamado my-first-project.Rproj en la carpeta asociada con el proyecto. Veremos cómo esto es útil más abajo.

Les dará opciones sobre dónde debe estar esta carpeta en su sistema de archivos. En este ejemplo, lo colocaremos en nuestra carpeta de inicio, aunque esto generalmente no es una buena práctica. Como describimos en la Sección 38.7 en el capítulo de Unix, quieren organizar su sistema de archivos siguiendo un enfoque jerárquico y con una carpeta llamada proyectos donde guardarán una carpeta para cada proyecto.
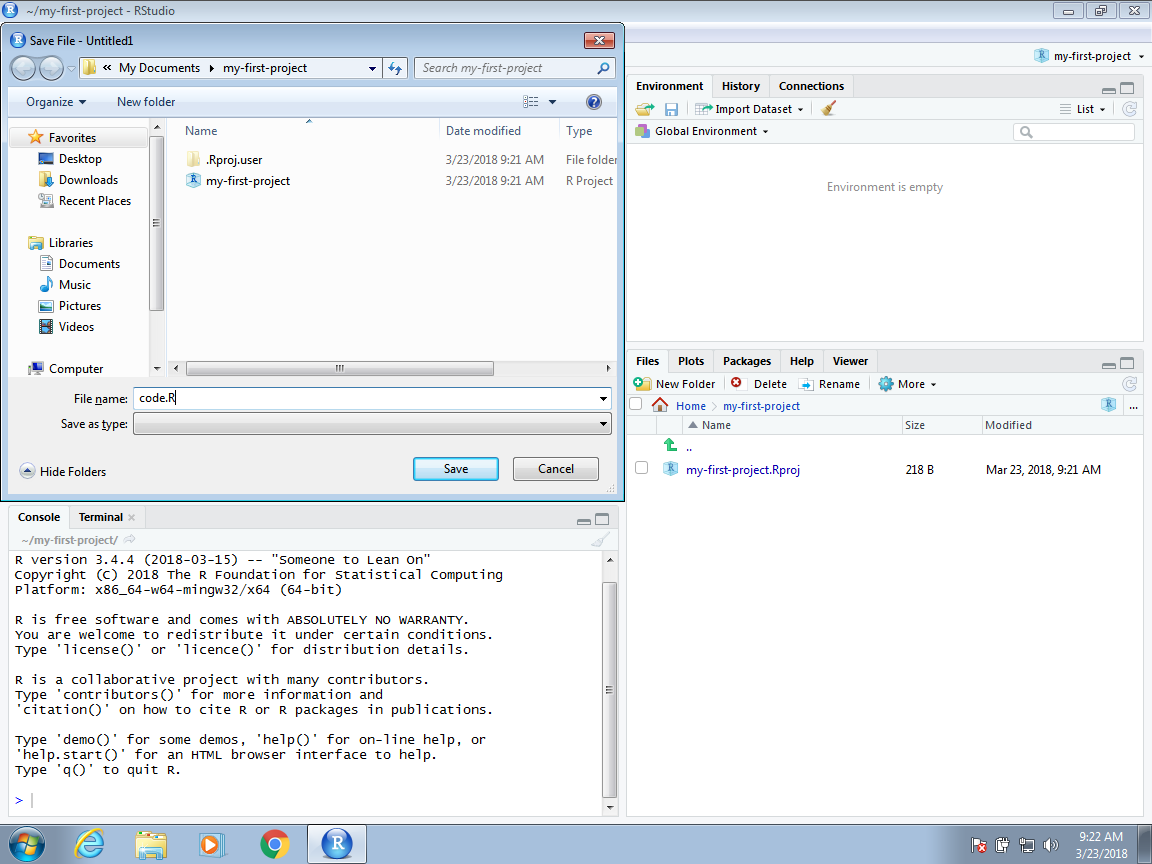
Cuando comiencen a usar RStudio con un proyecto, verán el nombre del proyecto en la esquina superior izquierda. Esto les recordará a qué proyecto pertenece esta sesión de RStudio en particular. Cuando abran una sesión de RStudio sin ningún proyecto, dirá Project:(None).
Al trabajar en un proyecto, todos los archivos se guardarán y se buscarán en la carpeta asociada con el proyecto. A continuación, mostramos un ejemplo de un script que escribimos y guardamos con el nombre code.R. Como usamos un nombre significativo para el proyecto, podemos ser un poco menos informativos cuando nombramos los archivos. Aunque no lo hacemos aquí, pueden tener varios scripts abiertos a la vez. Simplemente necesitan hacer clic en File, luego en New File y entonces elegir el tipo de archivo que desean editar.
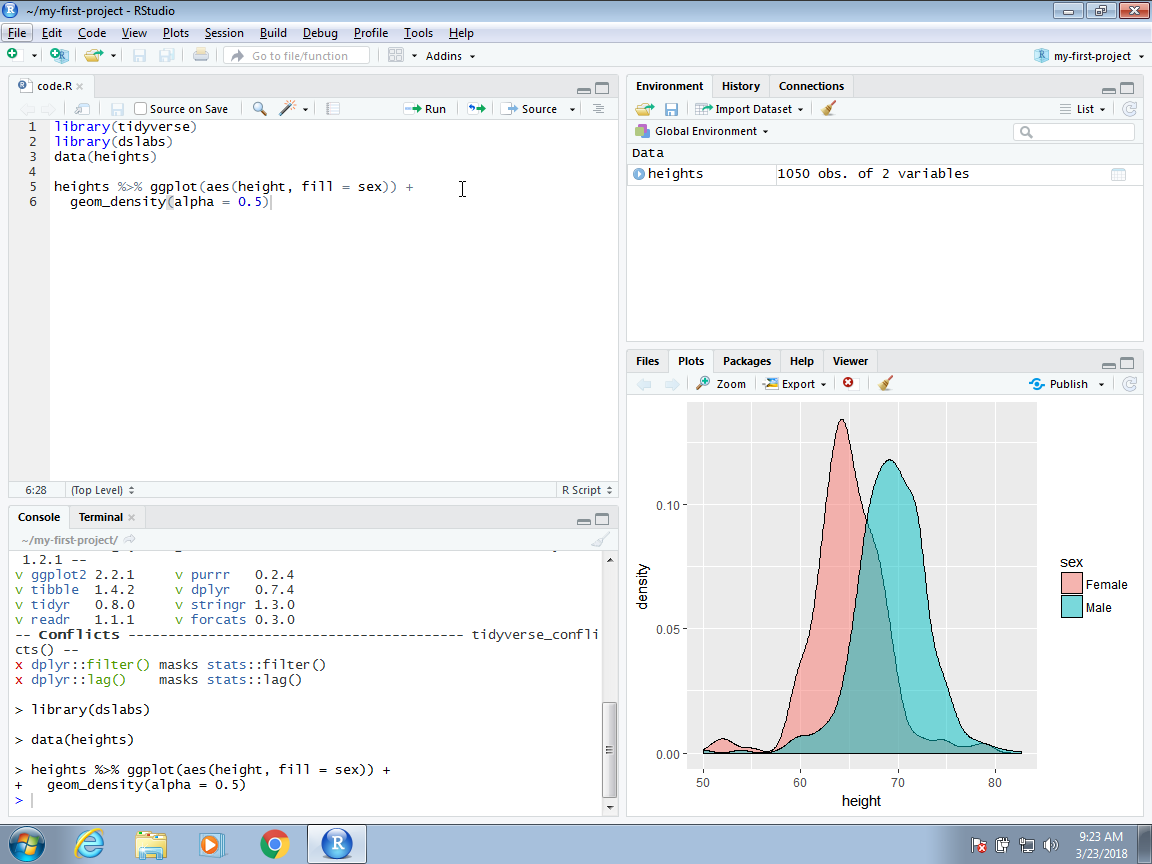
Una de las principales ventajas de usar Projects es que después de cerrar RStudio, si deseamos continuar donde pausamos, simplemente hacemos doble clic o abrimos el archivo guardado cuando creamos el proyecto de RStudio. En este caso, el archivo se llama my-first-project.Rproj. Si lo abrimos, RStudio se iniciará y abrirá los scripts que estábamos editando.
Otra ventaja es que si hacen clic en dos o más archivos diferentes de Rproj, iniciará nuevas sesiones de RStudio y R para cada uno.
40.2 R Markdown
R Markdown es un formato para documentos de programación literaria (literate programming en inglés). Se basa en markdown, un lenguaje de markup que se usa ampliamente para generar páginas HTML135. La programación literaria teje instrucciones, documentación y comentarios detallados entre el código ejecutable de la máquina, produciendo un documento que describe el programa que es mejor para la comprensión humana (Knuth 1984). A diferencia de un procesador de textos, como Microsoft Word, donde lo que ven es lo que obtienen, con R Markdown, necesitan compilar el documento en el informe final. El documento de R Markdown se ve diferente al producto final. De primer instancia, esto puede parecer una desventaja. Sin embargo, no lo es ya que en vez de producir gráficos e insertarlos uno por uno en el documento de procesamiento de texto, los gráficos se agregan automáticamente.
En RStudio, puede iniciar un documento de R Markdown haciendo clic en File, New File y entonces R Markdown. Luego se les pedirá que ingresen un título y un autor para su documento. Vamos a preparar un informe sobre asesinatos con armas de fuego, por lo que le daremos un nombre apropiado. También pueden escoger el formato del informe final: HTML, PDF o Microsoft Word. Más adelante, podemos cambiar esto fácilmente, pero aquí seleccionamos HTML ya que es el formato preferido para propósitos de depuración:
Esto generará un archivo de plantilla:
Como convención, usamos el sufijo Rmd para estos archivos.
Una vez que tengan más experiencia con R Markdown, podrán hacer esto sin la plantilla y simplemente comenzarán desde una plantilla en blanco.
En la plantilla, verán varias cosas para considerar.
40.2.1 El encabezado
En la parte superior ven:
---
title: "Report on Gun Murders"
author: "Rafael Irizarry"
date: "April 16, 2018"
output: html_document
---Todo lo que está entre --- es el encabezado. No necesitamos un encabezado, pero a menudo es útil. Pueden definir muchas otras cosas en el encabezado además de lo que se incluye en la plantilla. No discutimos esos aquí, pero hay mucha información disponible en línea. El único parámetro que destacaremos es output. Al cambiar esto a, por ejemplo, pdf_document, podemos controlar el tipo de output que se produce cuando compilamos.
40.2.2 Fragmentos de código R
En varios lugares del documento, vemos algo como lo siguiente:
```{r}
summary(pressure)
```Estos son fragmentos de código. Cuando compilan el documento, el código R dentro del fragmento, en este caso summary(pressure), será evaluado y el resultado incluido en esa posición en el documento final.
Para añadir sus propios fragmentos de R, pueden escribir los caracteres de arriba rápidamente con la combinación de teclas opción-I en Mac y Ctrl-Alt-I en Windows.
Esto aplica también a los gráficos; el gráfico se colocará en esa posición. Podemos escribir algo como esto:
```{r}
plot(pressure)
```Por defecto, el código también aparecerá. Para evitar esto, pueden usar el argumento echo=FALSE. Por ejemplo:
```{r echo=FALSE}
summary(pressure)
```Recomendamos acostumbrarse a añadir una etiqueta a los fragmentos de código R. Esto será muy útil al depurar, entre otras situaciones. Para hacer esto, agreguen una palabra descriptiva como esta:
```{r pressure-summary}
summary(pressure)
```40.2.3 Opciones globales
Uno de los fragmentos de R contiene una llamada que parece complicada:
```{r setup, include=FALSE}
knitr::opts_chunk$set(echo = TRUE)
```No cubriremos esto aquí, pero a medida que tengan más experiencia con R Markdown, aprenderán las ventajas de establecer opciones globales para el proceso de compilación.
40.2.4 knitR
Usamos el paquete knitR para compilar documentos de R Markdown. La función específica utilizada para compilar es knit, que toma un nombre de archivo como entrada. RStudio provee un botón que facilita la compilación del documento. En la siguiente captura de pantalla, hemos editado el documento para que se produzca un informe sobre asesinatos con armas de fuego. Pueden ver el archivo aquí: https://raw.githubusercontent.com/rairizarry/murders/master/report.Rmd. Ahora pueden hacer clic en el botón Knit:
La primera vez que hacen clic en el botón Knit, puede aparecer un cuadro de diálogo pidiéndoles que instalen los paquetes que necesitan.
Una vez que hayan instalado los paquetes, al hacer clic en Knit se compilará su archivo de R Markdown y emergerá el documento resultante.
Esto produce un documento HTML que pueden ver en su directorio de trabajo. Para verlo, abran un terminal y enumeren los archivos. Pueden abrir el archivo en un navegador y usarlo para presentar su análisis. Además, pueden producir un documento PDF o de Microsoft cambiando:
output: html_document a output: pdf_document o output: word_document.
También podemos producir documentos que se procesan en GitHub usando output: github_document.
Esto producirá un archivo de Markdown, con sufijo md, que se puede visualizar como una página de web en GitHub. Como hemos subido estos archivos a GitHub, pueden hacer clic en el archivo md y verán el informe:
Esta es una manera conveniente de compartir sus informes.
40.2.5 Más sobre R Markdown
Hay mucho más que pueden hacer con R Markdown. Le recomendamos que continúen aprendiendo a medida que adquieran más experiencia escribiendo informes en R. Hay muchos recursos gratuitos en el Internet que incluyen:
- Tutorial de RStudio: https://rmarkdown.rstudio.com
- La hoja de referencia: https://github.com/rstudio/cheatsheets/raw/master/translations/spanish/rmarkdown-2.0_Spanish.pdf
- El libro de knitR: https://yihui.name/knitr/
40.3 Organizando un proyecto de ciencia de datos
En esta sección, juntamos todo para crear el proyecto de asesinatos de EE.UU. y compartirlo en GitHub.
40.3.1 Crear directorios en Unix
En la Sección 38.7, demostramos cómo usar Unix para prepararnos para un proyecto de ciencia de datos usando un ejemplo. Aquí continuamos con este ejemplo y mostramos cómo usar RStudio. En la Sección 38.7, creamos los siguientes directorios usando Unix:
cd ~
cd projects
mkdir murders
cd murders
mkdir data rdas40.3.2 Crear un proyecto en RStudio
En la siguiente sección, crearemos un proyecto en RStudio. Primero vamos a File y luego a New Project… y, al ver las opciones, elegimos Existing Directory. Entonces escribimos la ruta completa del directorio murders creado anteriormente.
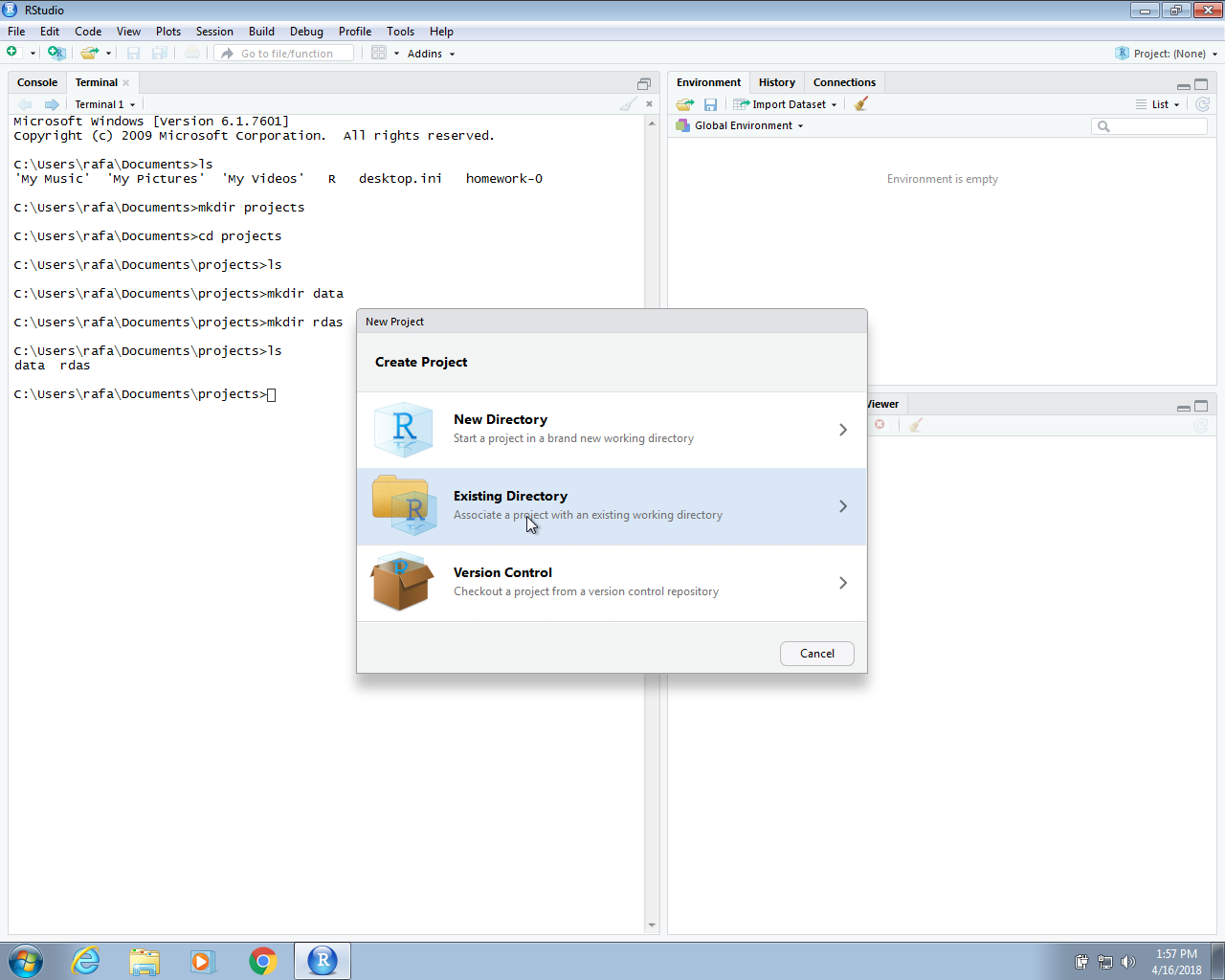
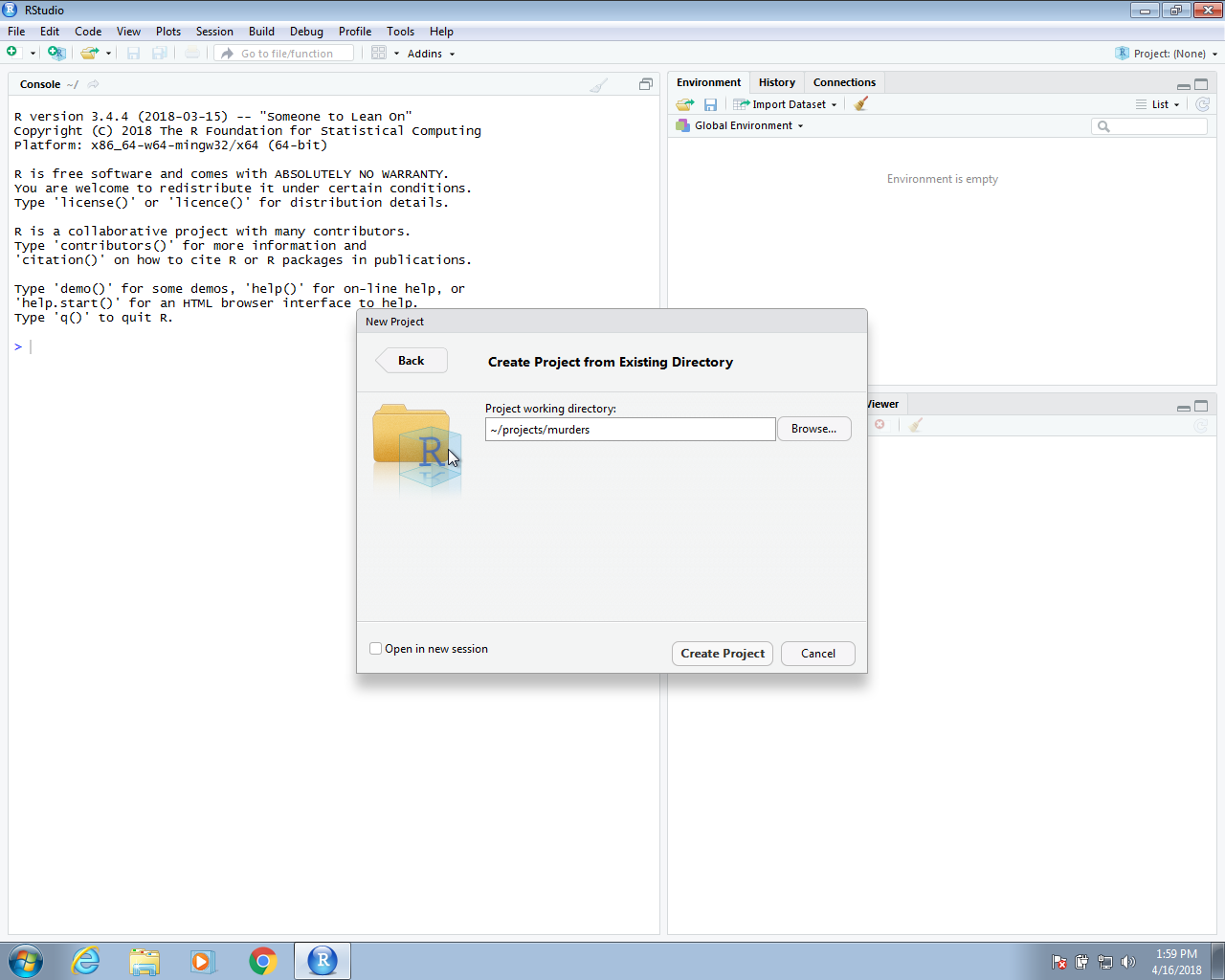
Una vez que hagan esto, verán los directorios rdas y data que crearon en la pestaña Files de RStudio.
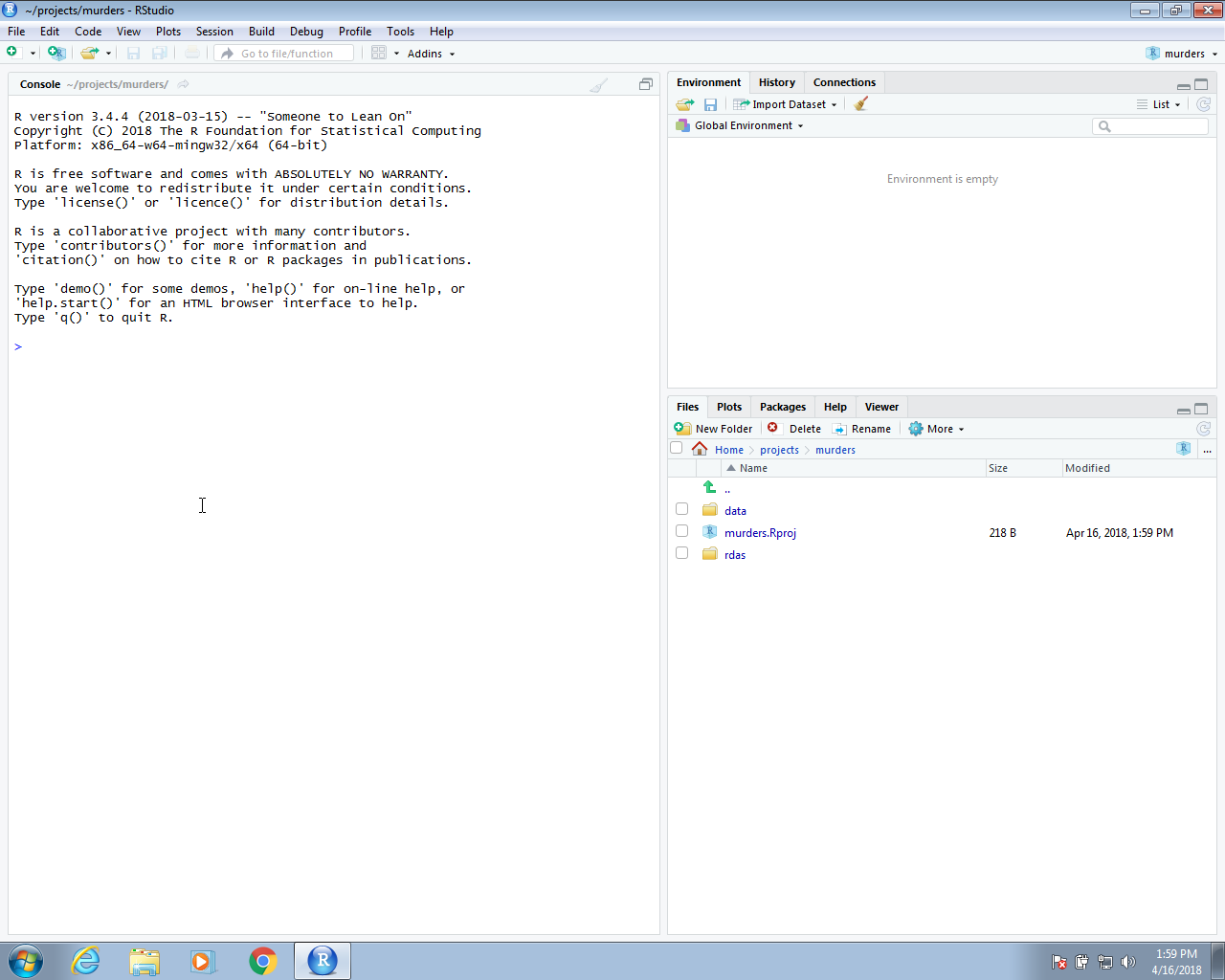
Tengan en cuenta que cuando estamos en este proyecto, nuestro directorio de trabajo predeterminado será ~/projects/murders. Pueden confirmar esto escribiendo getwd() en su sesión de R. Esto es importante porque nos ayudará a organizar el código cuando necesitemos escribir rutas de archivos. Consejo profesional: siempre usen rutas relativas en el código para proyectos de ciencia de datos. Estas deben ser relativas al directorio de trabajo que ha sido predeterminado. El problema con el uso de rutas completas es que es poco probable que sus códigos funcionen en sistemas de archivos distintos a los suyos, ya que las estructuras de directorio serán diferentes. Esto incluye el uso del directorio home ~ como parte de su ruta.
40.3.3 Editar algunos scripts de R
Ahora escribamos un script que descargue un archivo en el directorio de datos. Llamaremos a este archivo download-data.R.
El contenido de este archivo será:
url <- "https://raw.githubusercontent.com/rafalab/dslabs/master/inst/
extdata/murders.csv"
dest_file <- "data/murders.csv"
download.file(url, destfile = dest_file)Recuerden que estamos utilizando la ruta relativa data/murders.csv.
Ejecuten este código en R y verán que se agrega un archivo al directorio data.
Ahora estamos listos para escribir un script para leer estos datos y preparar una tabla que podamos usar para el análisis. Llamen al archivo wrangle-data.R. El contenido de este archivo será:
library(tidyverse)
murders <- read_csv("data/murders.csv")
murders <-murders |> mutate(region = factor(region),
rate = total/ population * 10^5)
save(murders, file = "rdas/murders.rda")Una vez más, tengan en cuenta que utilizamos rutas relativas exclusivamente.
En este archivo, presentamos un comando R que no hemos visto antes: save. El comando save guarda objetos en lo que se llama un archivo rda: rda es la abreviatura de datos de R. Recomendamos usar el sufijo .rda en archivos que guardan objetos R. Veran que .RData también se usa.
Si ejecutan el código anterior, el objeto de datos procesados se guardará en un archivo en el directorio rda. Aunque no es el caso aquí, este enfoque a menudo es práctico porque generar el objeto de datos que usamos para los análisis y gráficos finales puede ser un proceso complejo que requiere mucho tiempo. Entonces ejecutamos este proceso una vez y guardamos el archivo. Pero aún queremos poder generar el análisis completo a partir de los datos sin procesar.
Ahora estamos listos para escribir el archivo de análisis. Vamos a llamarlo analysis.R. El contenido debe ser el siguiente:
library(tidyverse)
load("rdas/murders.rda")
murders |> mutate(abb = reorder(abb, rate)) |>
ggplot(aes(abb, rate)) +
geom_bar(width = 0.5, stat = "identity", color = "black") +
coord_flip()Si ejecutan este análisis, verán que genera un gráfico.
40.3.4 Crear más directorios usando Unix
Ahora supongan que queremos guardar el gráfico generado para un informe o presentación. Podemos hacer esto con el comando ggsave de ggplot. ¿Pero dónde ponemos el gráfico? Deberíamos organizarnos sistemáticamente para que podamos guardar los gráficos en un directorio llamado figs. Comiencen creando un directorio escribiendo lo siguiente en el terminal:
mkdir figsy luego pueden agregar la línea:
ggsave("figs/barplot.png")a su script de R. Si ejecutan el script ahora, se guardará en un archivo png en el directorio figs. Si queremos copiar ese archivo a otro directorio donde estamos desarrollando una presentación, podemos evitar usar el mouse usando el comando cp en nuestro terminal.
40.3.5 Agregar un archivo README
Ahora tienen un análisis autónomo en un directorio. Una recomendación final es crear un archivo README.txt que describe lo que cada uno de estos archivos hace para el beneficio de otros que lean su código, incluyendo ustedes en el futuro. Esto no sería un script, sino solo algunas notas. Una de las opciones proporcionadas al abrir un nuevo archivo en RStudio es un archivo de texto. Puede guardar algo como esto en el archivo de texto:
We analyze US gun murder data collected by the FBI.
download-data.R - Downloads csv file to data directory
wrangle-data.R - Creates a derived dataset and saves as R object in rdas
directory
analysis.R - A plot is generated and saved in the figs directory.40.3.6 Inicializando un directorio Git
En la Sección 39.5, demostramos cómo inicializar un directorio Git y conectarlo al Upstream Repository en GitHub, que ya creamos en esa sección.
Podemos hacer esto en el terminal de Unix:
cd ~/projects/murders
git init
git add README.txt
git commit -m "First commit. Adding README.txt file just to get started"
git remote add origin `https://github.com/rairizarry/murders.git`
git push40.3.7 Add, commit y push archivos con RStudio
Podemos continuar adding y committing cada archivo, pero podría ser más fácil usar RStudio. Para hacer esto, inicien el proyecto abriendo el archivo Rproj. Deben aparecer los iconos git y pueden add, commit y push con estos.
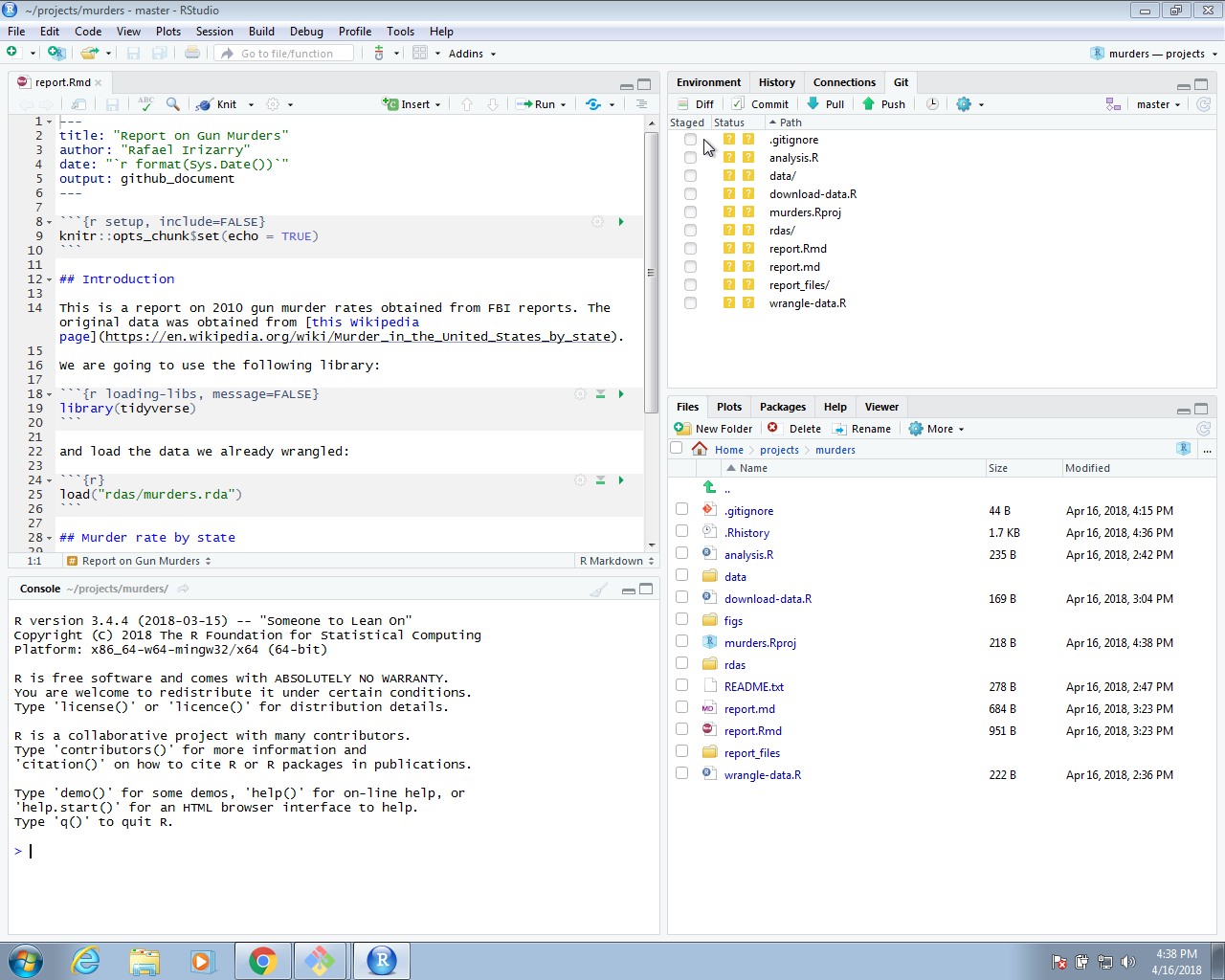
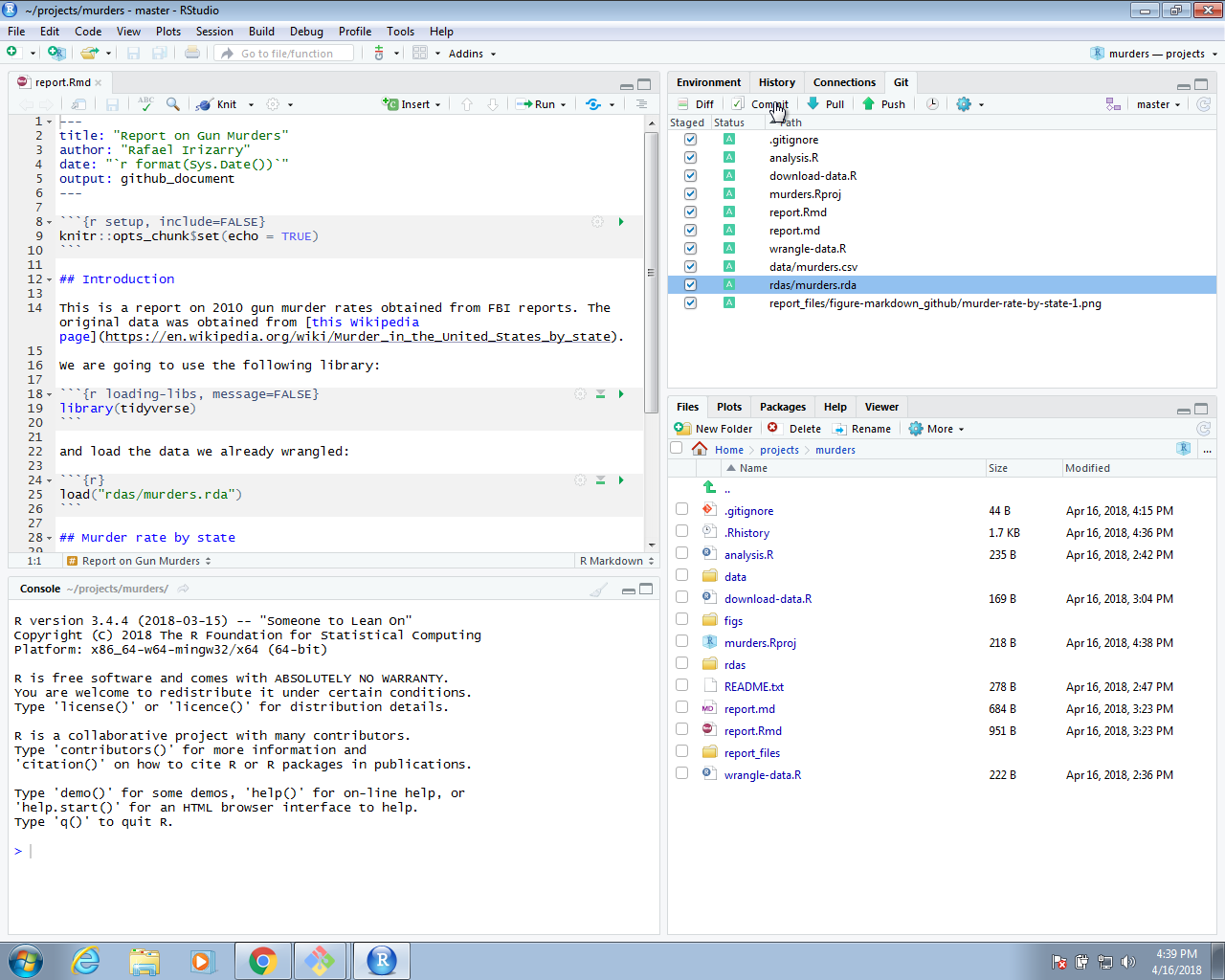
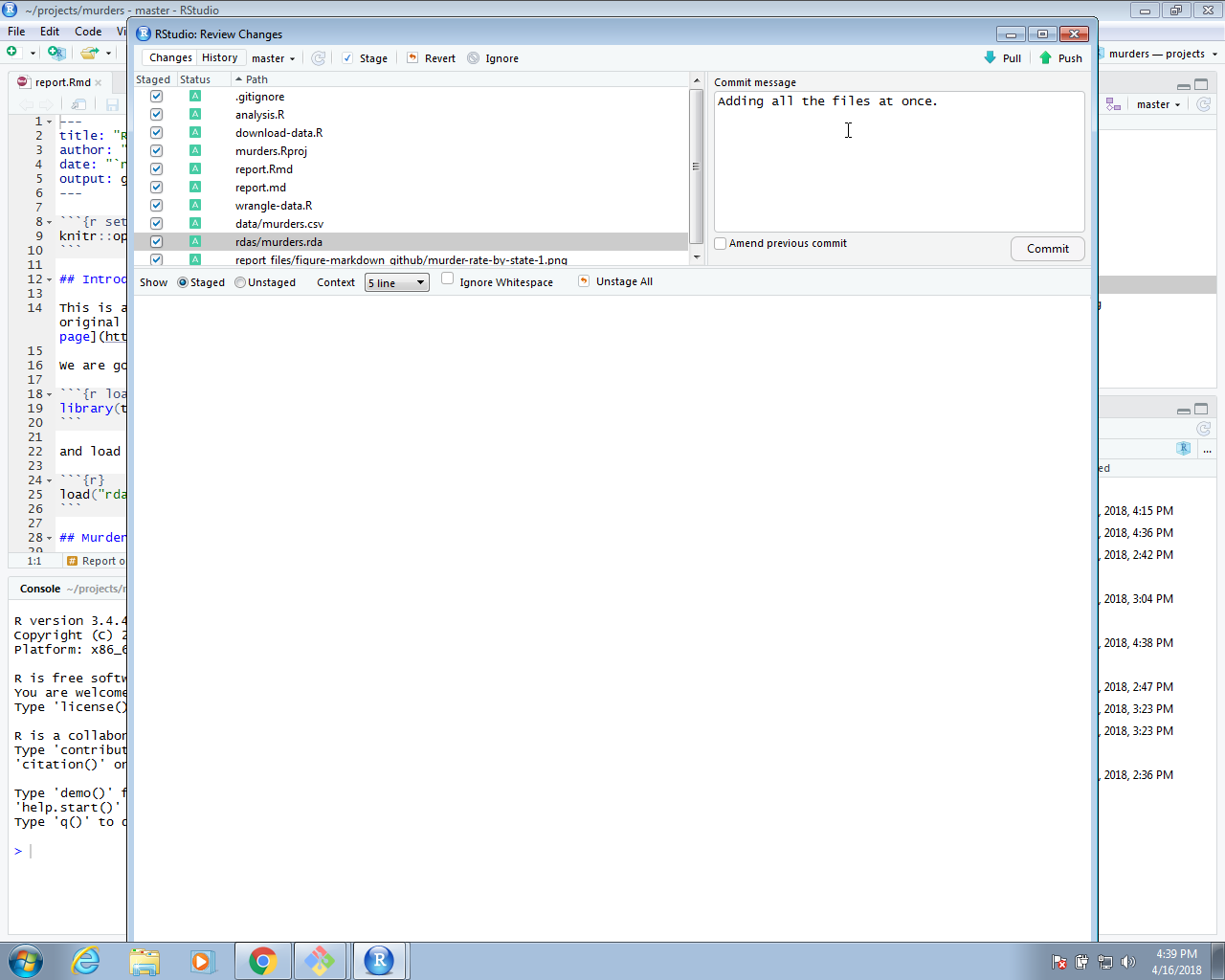
Ahora podemos ir a GitHub y confirmar que nuestros archivos están allí.
Pueden ver una versión de este proyecto, organizada con directorios de Unix, en GitHub136.
Pueden descargar una copia a su computadora utilizando el comando git clone en su terminal. Este comando creará un directorio llamado murders en su directorio de trabajo, así que tengan cuidado desde dónde lo llama.
git clone https://github.com/rairizarry/murders.git